Artigo escrito com a colaboração de Lara Reis
O pacote gtsummary apresenta tabelas analíticas e de resumo, prontas para a publicação.
O nome gtsummary surgiu em razão da intenção dos criadores de aproveitar todos os recursos do pacote gt. E a motivação por trás da criação do pacote veio do contato que os desenvolvedores tinham com o trabalho em bioestatistica. Todos os dias eles resumiam conjuntos de dados e modelos de regressão em R, que eventualmente eram incluídos em publicações.
Eles tinham seus próprios scripts para criação de tabelas específicas, mas, mesmo assim, muitas vezes precisavam modificar a formatação em um editor de documentos posteriormente, o que não levava a resultados reproduzíveis.
Instalação do pacote gtsummary
install.packages("gtsummary")
library(gtsummary)Dados
AVC %>% DT::datatable()Show 10 entries
| ID | Idade | Sexo | Altura | Peso | IMC | Classificação.IMC | Doença.crônica | Tempo.de.diagnóstico | Diagnóstico.Funcional | PIMáx | PEMáx | MIF | Classificação.MIF | EAT.10 | Disfagia | DASI | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 1 | 70 | Feminino | 140 | 60 | 21.42 | Baixo Peso | Hipertensão Arterial Sistêmica e Diabetes Mellitus | 6 | Hemiparesia à esquerda | 50 | 80 | 125 | Independência completa | 0 | 0 | 4.64 |
| 2 | 2 | 63 | Masculino | 163 | 80 | 24.53 | Adequado ou Eutrófico | Hipertensão Arterial Sistêmica | 7 | Hemiparesia à direita | 60 | 160 | 125 | Independência completa | 4 | 1 | 5.07 |
| 3 | 3 | 63 | Masculino | 165 | 72 | 21.81 | Baixo Peso | Hipertensão Arterial Sistêmica | 12 | Hemiparesia à esquerda | 50 | 100 | 119 | Independência completa | 0 | 0 | 5.07 |
| 4 | 4 | 64 | Feminino | 149 | 61 | 20.46 | Baixo Peso | Hipertensão Arterial Sistêmica | 48 | Hemiparesia à direita | 50 | 70 | 126 | Independência completa | 0 | 0 | 5.07 |
| 5 | 5 | 70 | Feminino | 155 | 58 | 18.7 | Baixo Peso | Hipertensão Arterial Sistêmica | 6 | Hemiparesia à direita | 50 | 50 | 115 | Independência completa | 0 | 0 | 4.64 |
| 6 | 6 | 68 | Masculino | 163 | 78 | 23.92 | Adequado ou Eutrófico | Hipertensão Arterial Sistêmica | 6 | Hemiparesia à esquerda | 80 | 110 | 123 | Independência completa | 0 | 0 | 3.3 |
| 7 | 7 | 67 | Feminino | 160 | 78.3 | 24.4 | Adequado ou Eutrófico | Hipertensão Arterial Sistêmica | 132 | Hemiparesia à esquerda | 60 | 70 | 124 | Independência completa | 0 | 0 | 5.5 |
| 8 | 8 | 73 | Masculino | 155 | 68.5 | 22.8 | Adequado ou Eutrófico | Não | 8 | Hemiparesia à esquerda | 40 | 50 | 123 | Independência completa | 0 | 0 | 3.3 |
| 9 | 9 | 71 | Masculino | 165 | 107 | 32.42 | Sobrepeso | Não | 6 | Hemiparesia à esquerda | 60 | 100 | 126 | Independência completa | 0 | 0 | 4.64 |
| 10 | 10 | 60 | Feminino | 160 | 60 | 18.75 | Baixo Peso | Hipertensão Arterial Sistêmica | 12 | Hemiparesia à esquerda | 80 | 70 | 126 | Independência completa | 0 | 0 | 4.64 |
Showing 1 to 10 of 61 entries
Funções do gtsummary
O pacote apresenta as seguintes funções principais:
- tbl_summary( ): calcula estatísticas descritivas e apresenta os resultados em tabelas resumo;
- tbl_cross( ): compara duas variáveis categóricas;
- tbl_uvregression( ): realiza uma regressão univariada e retorna uma tabela formatada com as principais estatísticas do modelo;
- tbl_regression( ): através de um objeto de um modelo de regressão multivariada retorna uma tabela formatada com as principais estatísticas do modelo;
- inline_text( ): possibilita a criação de relatórios reproduzíveis, relatando os resultados das tabelas no texto de um relatório de Markdown.
tbl_summary()
Uso básico
Por padrão:
- Os tipos de variáveis são identificadas e as estatísticas apropriadas são calculadas.
- Os atributos de cada variável do conjunto de dados são impressos automaticamente.
- Os níveis de variáveis são recuados e notas de rodapé são adicionadas.
AVC %>% tbl_summary()| Characteristic | N = 611 |
|---|---|
| ID | 31 (16, 46) |
| Idade | 68 (63, 73) |
| Sexo | |
| Feminino | 25 (41%) |
| Masculino | 36 (59%) |
| Altura | 160 (155, 165) |
| Peso | 69 (64, 78) |
| IMC | 24.7 (21.8, 26.9) |
| Classificação.IMC | |
| Adequado ou Eutrófico | 30 (49%) |
| Baixo Peso | 16 (26%) |
| Sobrepeso | 15 (25%) |
| Doença.crônica | |
| Hipertensão Arterial Sistêmica | 37 (61%) |
| Hipertensão Arterial Sistêmica e Diabetes Mellitus | 15 (25%) |
| Hipertensão Arterial Sistêmica e Insuficiência Renal | 1 (1.6%) |
| Não | 8 (13%) |
| Tempo.de.diagnóstico | 15 (9, 36) |
| Diagnóstico.Funcional | |
| Hemiparesia à direita | 25 (41%) |
| Hemiparesia à esquerda | 36 (59%) |
| PIMáx | 60 (45, 70) |
| PEMáx | 70 (50, 80) |
| MIF | 118 (95, 125) |
| Classificação.MIF | |
| Dependência modificada até 25% | 13 (21%) |
| Dependência modificada até 50% | 6 (9.8%) |
| Independência completa | 42 (69%) |
| EAT.10 | 0 (0, 3) |
| Disfagia | 16 (26%) |
| DASI | 4.47 (3.30, 4.98) |
| Unknown | 31 |
| 1 Statistics presented: Median (IQR); n (%) |
É possível personalizar a saída para ter tabelas mais interessantes
- Funções do gtsummary para modificar a aparência da tabela
| Argumento | Descrição |
|---|---|
| label | especificar as etiquetas das variáveis impressas na tabela |
| type | especifique o tipo de variável (por exemplo, contínua, categórica, etc.) |
| by | uma variável na qual as estatisticas resumo serão calculadas separadamente para cada nível |
| statistic | alterar as estatísticas de resumo apresentadas |
| digits | número de dígitos que as estatísticas de resumo serão arredondadas |
| missing | se deve exibir uma linha com o número de observações ausentes |
| percent | imprimir porcentagem de coluna, linha ou célula |
- Funções do gtsummary para adicionar informação à tabela
| Função | Descrição |
|---|---|
| add_p() | adicione valores p à saída comparando valores entre grupos |
| add_overall() | adicione uma coluna com estatísticas gerais de resumo |
| add_n() | adicione uma coluna com N (ou N faltando) para cada variável |
| add_stat_label() | adicionar etiqueta para as estatísticas de resumo mostradas em cada linha |
- Funções para formatar a tabela
| Função | Descrição |
|---|---|
| modify_header() | atualizar cabeçalhos de coluna |
| modify_footnote() | atualizar cabeçalhos abrangentes |
| bold_labels() | rótulos de variáveis em negrito |
| bold_levels() | níveis variáveis em negrito |
| italicize_labels() | itálico rótulos de variáveis |
| italicize_levels() | itálico níveis variáveis |
| bold_p() | valores p significativos em negrito |
Uso personalizado
- Análise descritiva para variáveis numéricas
t1 <-
AVC %>%
select(Idade, Altura, IMC, Peso,PIMáx, PEMáx, MIF, DASI ) %>%
tbl_summary(statistic = all_continuous() ~ "{mean}", missing = "no",
digits = list(all_continuous()~ 2)) %>%
modify_header(stat_0 ~ "**Média**") %>% add_n()
t2 <-
AVC %>%
select(Idade, Altura, IMC, Peso,PIMáx, PEMáx, MIF, DASI ) %>%
tbl_summary(statistic = all_continuous() ~ "{sd}", missing = "no",
digits = list(all_continuous()~ 2)) %>%
modify_header(stat_0 ~ "**S.D.**")
t3 <-
AVC %>%
select(Idade, Altura, IMC, Peso,PIMáx, PEMáx, MIF, DASI ) %>%
tbl_summary(statistic = all_continuous() ~ "{min}", missing = "no",
digits = list(all_continuous()~ 2)) %>%
modify_header(stat_0 ~ "**Mín**")
t4 <-
AVC %>%
select(Idade, Altura, IMC, Peso,PIMáx, PEMáx, MIF, DASI ) %>%
tbl_summary(statistic = all_continuous() ~ "{p25}", missing = "no",
digits = list(all_continuous()~ 2)) %>%
modify_header(stat_0 ~ "**1ºQ**")
t5 <-
AVC %>%
select(Idade, Altura, IMC, Peso,PIMáx, PEMáx, MIF, DASI ) %>%
tbl_summary(statistic = all_continuous() ~ "{p50}", missing = "no",
digits = list(all_continuous()~ 2)) %>%
modify_header(stat_0 ~ "**2ºQ**")
t6 <-
AVC %>%
select(Idade, Altura, IMC, Peso,PIMáx, PEMáx, MIF, DASI ) %>%
tbl_summary(statistic = all_continuous() ~ "{p75}", missing = "no",
digits = list(all_continuous()~ 2)) %>%
modify_header(stat_0 ~ "**3ºQ**")
t7 <-
AVC %>%
select(Idade, Altura, IMC, Peso,PIMáx, PEMáx, MIF, DASI ) %>%
tbl_summary(statistic = all_continuous() ~ "{max}", missing = "no",
digits = list(all_continuous()~ 2)) %>%
modify_header(stat_0 ~ "**Máx.**")
tbl_numerica <- tbl_merge(list(t1, t2,t3,t4,t5,t6,t7)) %>%
modify_header(update = list(label ~ "**Variáveis**")) %>% #modificando o nome da coluna
modify_footnote(everything() ~ NA_character_) %>% #retiradando os significados das estatisticas
modify_spanning_header(everything() ~ NA_character_) #retirando o nome da tabela
tbl_numerica| Variáveis | N | Média | S.D. | Mín | 1ºQ | 2ºQ | 3ºQ | Máx. |
|---|---|---|---|---|---|---|---|---|
| Idade | 61 | 69.05 | 7.32 | 60.00 | 63.00 | 68.00 | 73.00 | 87.00 |
| Altura | 61 | 160.57 | 9.17 | 140.00 | 155.00 | 160.00 | 165.00 | 184.00 |
| IMC | 61 | 24.75 | 4.71 | 15.00 | 21.81 | 24.71 | 26.90 | 41.90 |
| Peso | 61 | 70.61 | 10.71 | 43.50 | 64.00 | 69.40 | 77.60 | 107.00 |
| PIMáx | 61 | 58.77 | 17.29 | 25.00 | 45.00 | 60.00 | 70.00 | 100.00 |
| PEMáx | 61 | 70.00 | 23.66 | 20.00 | 50.00 | 70.00 | 80.00 | 160.00 |
| MIF | 61 | 108.13 | 21.37 | 50.00 | 95.00 | 118.00 | 125.00 | 126.00 |
| DASI | 30 | 4.30 | 1.06 | 2.74 | 3.30 | 4.47 | 4.98 | 6.70 |
- Análise descritiva para variáveis categóricas
categ <- AVC %>%
select(Sexo, Disfagia,Classificação.IMC, Doença.crônica,Diagnóstico.Funcional, Classificação.MIF, ) %>%
tbl_summary(
type = all_categorical() ~ "categorical") %>%
modify_header(update =list(label ~ "**Variáveis**"))%>%
bold_labels() #níveis variáveis em negrito
categ| Variáveis | N = 611 |
|---|---|
| Sexo | |
| Feminino | 25 (41%) |
| Masculino | 36 (59%) |
| Disfagia | |
| 0 | 45 (74%) |
| 1 | 16 (26%) |
| Classificação.IMC | |
| Adequado ou Eutrófico | 30 (49%) |
| Baixo Peso | 16 (26%) |
| Sobrepeso | 15 (25%) |
| Doença.crônica | |
| Hipertensão Arterial Sistêmica | 37 (61%) |
| Hipertensão Arterial Sistêmica e Diabetes Mellitus | 15 (25%) |
| Hipertensão Arterial Sistêmica e Insuficiência Renal | 1 (1.6%) |
| Não | 8 (13%) |
| Diagnóstico.Funcional | |
| Hemiparesia à direita | 25 (41%) |
| Hemiparesia à esquerda | 36 (59%) |
| Classificação.MIF | |
| Dependência modificada até 25% | 13 (21%) |
| Dependência modificada até 50% | 6 (9.8%) |
| Independência completa | 42 (69%) |
| 1 Statistics presented: n (%) |
- Tabela de contingência
AVC %>%
select(Disfagia, Sexo, Classificação.IMC,Doença.crônica, Diagnóstico.Funcional,
Classificação.MIF,) %>%
mutate(Disfagia = factor(Disfagia, labels = c("Sem risco", "Com Risco"))) %>%
tbl_summary(
by = Disfagia,
missing = "no",
) %>%
add_p(pvalue_fun = ~style_pvalue(.x, digits = 3)) %>%
modify_header(update =list(label ~ "**Variáveis**", p.value ~ "**Valor-p**"))%>%
bold_labels() %>%
bold_p(t = 0.05)| Variáveis | Sem risco, N = 451 | Com Risco, N = 161 | Valor-p2 |
|---|---|---|---|
| Sexo | 0.973 | ||
| Feminino | 19 (42%) | 6 (38%) | |
| Masculino | 26 (58%) | 10 (62%) | |
| Classificação.IMC | 0.098 | ||
| Adequado ou Eutrófico | 19 (42%) | 11 (69%) | |
| Baixo Peso | 12 (27%) | 4 (25%) | |
| Sobrepeso | 14 (31%) | 1 (6.2%) | |
| Doença.crônica | 0.782 | ||
| Hipertensão Arterial Sistêmica | 26 (58%) | 11 (69%) | |
| Hipertensão Arterial Sistêmica e Diabetes Mellitus | 11 (24%) | 4 (25%) | |
| Hipertensão Arterial Sistêmica e Insuficiência Renal | 1 (2.2%) | 0 (0%) | |
| Não | 7 (16%) | 1 (6.2%) | |
| Diagnóstico.Funcional | 0.531 | ||
| Hemiparesia à direita | 20 (44%) | 5 (31%) | |
| Hemiparesia à esquerda | 25 (56%) | 11 (69%) | |
| Classificação.MIF | 0.050 | ||
| Dependência modificada até 25% | 9 (20%) | 4 (25%) | |
| Dependência modificada até 50% | 2 (4.4%) | 4 (25%) | |
| Independência completa | 34 (76%) | 8 (50%) | |
| 1 Statistics presented: n (%) 2 Statistical tests performed: chi-square test of independence; Fisher’s exact test |
tbl_cross
- Adiciona automaticamente um cabeçalho com o nome ou rótulo da variável de comparação.
- Usa percent = “cell” por padrão.
- Adiciona totais de margem de linha e coluna (personalizável).
tc1 <- AVC %>%
tbl_cross(
row = Sexo,
col = Disfagia,
percent = "column"
) %>%
add_p()
tc1| DISFAGIA | DISFAGIA | |||
|---|---|---|---|---|
| Characteristic | 0 | 1 | Total | p-value1 |
| Sexo | >0.9 | |||
| Feminino | 19 (42%) | 6 (38%) | 25 (41%) | |
| Masculino | 26 (58%) | 10 (62%) | 36 (59%) | |
| Total | 45 (100%) | 16 (100%) | 61 (100%) | |
| 1 chi-square test of independence |
As funções tbl_uvregression() e tbl_regression() do gtsummary fornecem tabelas com as estatísticas resumo de um modelo de regressão. Elas apresentam as mesmas funções para personalização
- Argumentos para modificar a aparência.
| Argumento | Descrição |
|---|---|
| label | modificar rótulos de variáveis na tabela |
| exponentiate | exponenciar os coeficientes do modelo |
| include | nomes das variáveis a serem incluídas na saída. O padrão é todas as variáveis |
| show_single_row | Por padrão, as variáveis categóricas são impressas em várias linhas. Se uma variável é dicotômica e você deseja imprimir o coeficiente de regressão em uma única linha, inclua o nome da variável aqui. |
| conf.level | nível de confiança do intervalo de confiança |
| intercept | indica se incluir a interceptação |
| estimate_fun | função para arredondar e formatar estimativas de coeficiente |
| pvalue_fun | função para arredondar e formatar valores-p |
tbl_uvregression()
A tbl_uvregression() produz uma tabela de modelos de regressão univariada.
| Argumento | Descrição |
|---|---|
| method | método de regressão |
| y | variável resposta |
| x | variáveis explicativas. Por padrão, todas as outras são selecionadas |
| method.args | lista de argumentos da regressão |
Uso básico
AVC %>%
select(Disfagia,Idade, Altura,Peso, IMC, Tempo.de.diagnóstico, PIMáx, PEMáx, MIF, DASI) %>%
tbl_uvregression(
method = glm,
y = Disfagia,
method.args = list(family = binomial),
exponentiate = TRUE,
)| Characteristic | N | OR1 | 95% CI1 | p-value |
|---|---|---|---|---|
| Idade | 61 | 1.08 | 0.99, 1.17 | 0.072 |
| Altura | 61 | 1.06 | 1.00, 1.14 | 0.078 |
| Peso | 61 | 0.99 | 0.93, 1.04 | 0.7 |
| IMC | 61 | 0.97 | 0.85, 1.10 | 0.6 |
| Tempo.de.diagnóstico | 61 | 0.99 | 0.96, 1.01 | 0.3 |
| PIMáx | 61 | 0.94 | 0.89, 0.98 | 0.006 |
| PEMáx | 61 | 0.98 | 0.95, 1.01 | 0.2 |
| MIF | 61 | 0.97 | 0.94, 1.00 | 0.031 |
| DASI | 30 | 0.62 | 0.20, 1.60 | 0.4 |
| 1 OR = Odds Ratio, CI = Confidence Interval |
Uso personalizado
AVC %>%
select(Disfagia,Idade, Altura,Peso, IMC, Tempo.de.diagnóstico, PIMáx, PEMáx, MIF, DASI) %>%
tbl_uvregression(
method = glm,
y = Disfagia,
method.args = list(family = binomial),
exponentiate = TRUE,
pvalue_fun = ~style_pvalue(.x, digits = 2))%>%
bold_p() %>%
modify_header(
update =list(label ~ "**Variáveis**" ,
p.value ~ "**Valor-p**",
ci ~ "**I.C. 95%**"))| Variáveis | N | OR1 | I.C. 95%1 | Valor-p |
|---|---|---|---|---|
| Idade | 61 | 1.08 | 0.99, 1.17 | 0.072 |
| Altura | 61 | 1.06 | 1.00, 1.14 | 0.078 |
| Peso | 61 | 0.99 | 0.93, 1.04 | 0.70 |
| IMC | 61 | 0.97 | 0.85, 1.10 | 0.64 |
| Tempo.de.diagnóstico | 61 | 0.99 | 0.96, 1.01 | 0.27 |
| PIMáx | 61 | 0.94 | 0.89, 0.98 | 0.006 |
| PEMáx | 61 | 0.98 | 0.95, 1.01 | 0.22 |
| MIF | 61 | 0.97 | 0.94, 1.00 | 0.031 |
| DASI | 30 | 0.62 | 0.20, 1.60 | 0.35 |
| 1 OR = Odds Ratio, CI = Confidence Interval |
tbl_regression()
A tbl_regression() produz uma tabela de modelos de regressão multivariada.
Uso básico
modelo_mult <- lm(MIF ~ Idade + Peso + Altura + Sexo +
Classificação.IMC + Doença.crônica+ Disfagia+
Tempo.de.diagnóstico + Diagnóstico.Funcional +
PIMáx + PEMáx ,
AVC)
r_multi <- modelo_mult %>%
tbl_regression()
r_multi| Characteristic | Beta | 95% CI1 | p-value |
|---|---|---|---|
| Idade | -1.3 | -2.1, -0.45 | 0.003 |
| Peso | 0.39 | -0.36, 1.1 | 0.3 |
| Altura | -0.10 | -0.78, 0.58 | 0.8 |
| Sexo | |||
| Feminino | — | — | |
| Masculino | 9.9 | -2.4, 22 | 0.11 |
| Classificação.IMC | |||
| Adequado ou Eutrófico | — | — | |
| Baixo Peso | 4.1 | -10, 18 | 0.6 |
| Sobrepeso | -9.1 | -24, 5.6 | 0.2 |
| Doença.crônica | |||
| Hipertensão Arterial Sistêmica | — | — | |
| Hipertensão Arterial Sistêmica e Diabetes Mellitus | 2.1 | -10, 14 | 0.7 |
| Hipertensão Arterial Sistêmica e Insuficiência Renal | 18 | -23, 58 | 0.4 |
| Não | 3.3 | -14, 21 | 0.7 |
| Disfagia | -8.9 | -22, 4.0 | 0.2 |
| Tempo.de.diagnóstico | 0.05 | -0.09, 0.19 | 0.5 |
| Diagnóstico.Funcional | |||
| Hemiparesia à direita | — | — | |
| Hemiparesia à esquerda | 0.24 | -10.0, 10 | >0.9 |
| PIMáx | 0.00 | -0.38, 0.38 | >0.9 |
| PEMáx | 0.06 | -0.22, 0.34 | 0.7 |
| 1 CI = Confidence Interval |
Uso personalizado
#regresão linear
linear <- lm(PEMáx ~ Sexo +Classificação.IMC + Doença.crônica
+ Diagnóstico.Funcional +Disfagia + Peso+
Idade + Altura + IMC+
Tempo.de.diagnóstico +
PIMáx + MIF+ DASI, AVC)
inicial <- linear %>%
tbl_regression(pvalue_fun = ~ style_pvalue ( .x , digits = 3))%>%
modify_header(update =list(label ~ "**Variáveis**",p.value ~ "**Valor-p**",
ci ~ "**I.C. 95%**")) %>%
bold_labels() %>% #grifa a variavel
bold_p(t = 0.05) #grifa os p valores menores que t
inicial| Variáveis | Beta | I.C. 95%1 | Valor-p |
|---|---|---|---|
| Sexo | |||
| Feminino | — | — | |
| Masculino | 28 | -8.2, 63 | 0.119 |
| Classificação.IMC | |||
| Adequado ou Eutrófico | — | — | |
| Baixo Peso | 13 | -34, 60 | 0.552 |
| Sobrepeso | -8.0 | -71, 55 | 0.787 |
| Doença.crônica | |||
| Hipertensão Arterial Sistêmica | — | — | |
| Hipertensão Arterial Sistêmica e Diabetes Mellitus | 0.03 | -36, 36 | 0.999 |
| Hipertensão Arterial Sistêmica e Insuficiência Renal | 6.4 | -69, 82 | 0.857 |
| Não | -11 | -70, 47 | 0.689 |
| Diagnóstico.Funcional | |||
| Hemiparesia à direita | — | — | |
| Hemiparesia à esquerda | 2.7 | -28, 34 | 0.855 |
| Disfagia | -10 | -51, 31 | 0.603 |
| Peso | -3.7 | -13, 5.3 | 0.386 |
| Idade | -1.9 | -4.7, 1.0 | 0.177 |
| Altura | 0.89 | -3.1, 4.8 | 0.636 |
| IMC | 16 | -13, 45 | 0.255 |
| Tempo.de.diagnóstico | -0.04 | -0.35, 0.28 | 0.803 |
| PIMáx | 0.49 | -0.32, 1.3 | 0.212 |
| MIF | 0.21 | -1.3, 1.7 | 0.770 |
| DASI | 0.89 | -19, 21 | 0.926 |
| 1 CI = Confidence Interval |
Usando novamente a função tbl_merge() para juntar duas tabelas, podemos chamá-las de modelo inicial e modelo final
linear2 <- lm(PEMáx ~ Sexo +
Idade +
PIMáx, AVC)
final <- linear2 %>% tbl_regression(pvalue_fun = ~ style_pvalue(.x , digits = 3)) %>%
modify_header(update =list(label ~ "**Variáveis**" ,
p.value ~ "**Valor-p**")) %>%
bold_labels() %>% #grifa a variavel
bold_p(t = 0.05) %>% #grifa os p valores menores que t
modify_header(update =list(label ~ "**Variáveis**"))
#Podemos juntar duas tabelas
tbl_merge_ex2 <-
tbl_merge(
tbls = list(inicial, final),
tab_spanner = c("**Modelo inicial**", "**Modelo final**")
)
tbl_merge_ex2| BETA | I.C. 95%1 | VALOR-P | BETA | 95% CI1 | ||
|---|---|---|---|---|---|---|
| Sexo | ||||||
| Feminino | — | — | — | — | ||
| Masculino | 28 | -8.2, 63 | 0.119 | 11 | 0.51, 21 | 0.040 |
| Classificação.IMC | ||||||
| Adequado ou Eutrófico | — | — | ||||
| Baixo Peso | 13 | -34, 60 | 0.552 | |||
| Sobrepeso | -8.0 | -71, 55 | 0.787 | |||
| Doença.crônica | ||||||
| Hipertensão Arterial Sistêmica | — | — | ||||
| Hipertensão Arterial Sistêmica e Diabetes Mellitus | 0.03 | -36, 36 | 0.999 | |||
| Hipertensão Arterial Sistêmica e Insuficiência Renal | 6.4 | -69, 82 | 0.857 | |||
| Não | -11 | -70, 47 | 0.689 | |||
| Diagnóstico.Funcional | ||||||
| Hemiparesia à direita | — | — | ||||
| Hemiparesia à esquerda | 2.7 | -28, 34 | 0.855 | |||
| Disfagia | -10 | -51, 31 | 0.603 | |||
| Peso | -3.7 | -13, 5.3 | 0.386 | |||
| Idade | -1.9 | -4.7, 1.0 | 0.177 | -0.90 | -1.6, -0.18 | 0.015 |
| Altura | 0.89 | -3.1, 4.8 | 0.636 | |||
| IMC | 16 | -13, 45 | 0.255 | |||
| Tempo.de.diagnóstico | -0.04 | -0.35, 0.28 | 0.803 | |||
| PIMáx | 0.49 | -0.32, 1.3 | 0.212 | 0.54 | 0.23, 0.86 | 0.001 |
| MIF | 0.21 | -1.3, 1.7 | 0.770 | |||
| DASI | 0.89 | -19, 21 | 0.926 | |||
| 1 CI = Confidence Interval |
inline_text()
Resultados de uma regressão de forma reproduzível
`r inline_text(tabela, variable = ,pattern = "{}")`Argumentos:
- nome da tabela
- variable: a variável que deseja ter os resultados
- pattern: as estatísticas que serão imprimidas entre chaves e aspas ({estimate},{conf.low},{conf.high},{p.value},{conf.level},{N})
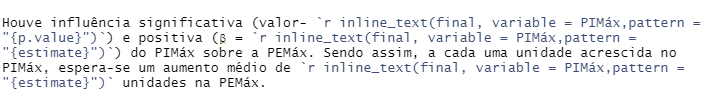
Houve influência significativa (valor- p=0.001) e positiva (β = 0.54) do PIMáx sobre a PEMáx. Sendo assim, a cada uma unidade acrescida no PIMáx, espera-se um aumento médio de 0.54 unidades na PEMáx.
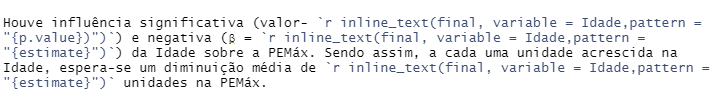
Houve influência significativa (valor- p=0.015)) e negativa (β = -0.90) da Idade sobre a PEMáx. Sendo assim, a cada uma unidade acrescida na Idade, espera-se um diminuição média de -0.90 unidades na PEMáx.
Resultados de uma análise descritiva de forma reproduzível
`r inline_text(tabela, variable = , column = ,level = ,pattern = "{}")`Argumentos:
- nome da tabela;
- variable: a variável que deseja ter os resultados;
- column: nível da variável escolhida (coluna);
- level: nível da variável categórica (linha);
- pattern: as estatísticas que devem ser imprimidas entre aspas e chaves( ).
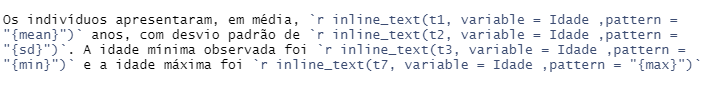
Os indivíduos apresentaram, em média, 69.05 anos, com desvio padrão de 7.32. A idade mínima observada foi 60.00 e a idade máxima foi 87.00

A maioria dos indivíduos (59%) é do sexo masculino.